AI mới của Meta dự đoán hình dạng của 600 triệu protein mà giới khoa học chưa hề biết đến
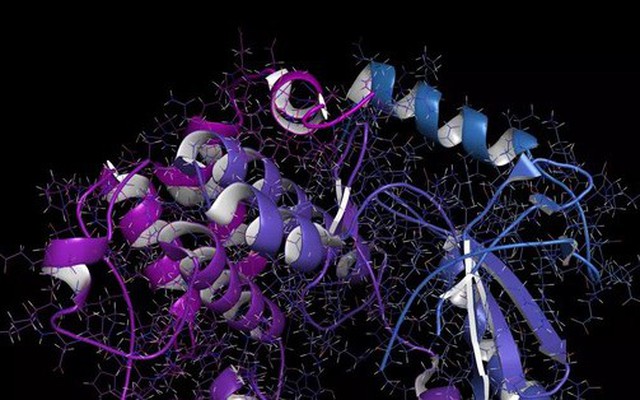
Một MEK1 hoặc protein kinase kinase 1(thỏ) được kích hoạt bằng mitogen.
Các nhà khoa học tại Meta, công ty mẹ của Facebook và Instagram, đã sử dụng mô hình ngôn ngữ trí tuệ nhân tạo (AI) để dự đoán cấu trúc chưa biết của hơn 600 triệu protein thuộc virus, vi khuẩn và các vi sinh vật khác.
- 18-11-2022Làm thế nào để tích hợp GPLX, thẻ BHYT vào tài khoản định danh điện tử?
- 18-11-2022Cách Colin Huang đưa Pinduoduo trở thành thế lực cạnh tranh với Alibaba và JD.com tại Trung Quốc
- 18-11-2022Giá Bitcoin hôm nay 18/11: Bitcoin giảm mạnh, thị trường tiền ảo ngập sắc đỏ
Chương trình có tên ESMFold, đã sử dụng một mô hình ban đầu được thiết kế để giải mã ngôn ngữ của con người nhằm đưa ra dự đoán chính xác về các vòng xoắn được thực hiện bởi các protein xác định cấu trúc 3D của chúng.
Các dự đoán này, đã được biên soạn trong ESM Metagenomic Atlas mã nguồn mở, có thể được sử dụng để giúp phát triển các loại thuốc mới, mô tả các chức năng của vi sinh vật chưa được biết đến và theo dõi các mối liên hệ tiến hóa giữa các loài có quan hệ họ hàng xa.
ESMFold không phải là chương trình đầu tiên đưa ra dự đoán về protein. Năm 2022, công ty DeepMind thuộc sở hữu của Google thông báo rằng chương trình dự đoán protein AlphaFold của họ đã giải mã hình dạng của khoảng 200 triệu protein mà khoa học biết đến.
Meta cho biết, ESMFold không chính xác bằng AlphaFold, nhưng nó nhanh hơn 60 lần so với chương trình của DeepMind. Kết quả vẫn chưa được bình duyệt.
Biết hình dạng của protein là cách tốt nhất để hiểu chức năng của nó, nhưng có một số cách đáng kinh ngạc mà sự kết hợp của các axit amin trong các trình tự khác nhau có thể hình thành.
Cách tiêu chuẩn vàng để xác định cấu trúc của protein là sử dụng tinh thể học tia X - xem cách các chùm ánh sáng năng lượng cao nhiễu xạ xung quanh protein - nhưng đây là một phương pháp tốn nhiều công sức có thể mất nhiều tháng hoặc nhiều năm để tạo ra kết quả và nó không hiệu quả cho tất cả các loại protein. Sau nhiều thập kỷ làm việc, hơn 100.000 cấu trúc protein đã được giải mã thông qua tinh thể học tia X.
Để tìm ra cách giải quyết vấn đề này, các nhà nghiên cứu Meta đã chuyển sang một mô hình máy tính phức tạp được thiết kế để giải mã và đưa ra dự đoán về ngôn ngữ của con người, đồng thời áp dụng mô hình này vào ngôn ngữ của chuỗi protein.
Để kiểm tra mô hình này, các nhà khoa học đã chuyển sang cơ sở dữ liệu AND hệ gien được lấy từ những nơi đa dạng như đất, nước biển, ruột và da của con người. Bằng cách cung cấp dữ liệu ADN vào chương trình ESMFold, các nhà nghiên cứu đã dự đoán cấu trúc của hơn 617 triệu protein chỉ trong hai tuần.
Con số này nhiều hơn 400 triệu so với AlphaFold tuyên bố đã giải mã cách đây 4 tháng, khi họ tuyên bố đã suy luận ra cấu trúc protein của hầu hết mọi loại protein được biết đến.
Điều này có nghĩa là nhiều loại protein này chưa từng được nhìn thấy trước đây, có thể là do chúng đến từ các sinh vật chưa được biết đến. Hơn 200 triệu dự đoán về protein của ESMFold được cho là có chất lượng cao, có nghĩa là chương trình đã có thể dự đoán các hình dạng với độ chính xác đến cấp độ nguyên tử.
Theo Live Science
Tiền Phong
